文章|2020年10分+纯生信文章带你领略Nature子刊之驱动突变文章套路( 二 )
Fig.1展示了通路和网络分析的流程图。利用多个通路/基因片段的数据库定义了一种非编码增值(NCVA)程序来确定那些非编码得分对编码和非编码分析结果有显著影响的基因,其中NCVA的结果增强了非编码数据的结果。并最终定义了一个共识程序,以结合重要的路径和网络识别这七个算法。编码变异的87个通路牵连驱动基因(PID-C)是大多数(≥4/7)编码数据方法报道的基因集合。93个具有非编码变异的通路牵连驱动基因(PID-N)是大多数方法在非编码数据或NCVA结果中报道的一组基因。只有5个基因(CTNNB1, DDX3X, SF3B1, TGFBR2, and TP53)同时是PID-C和PID-N基因。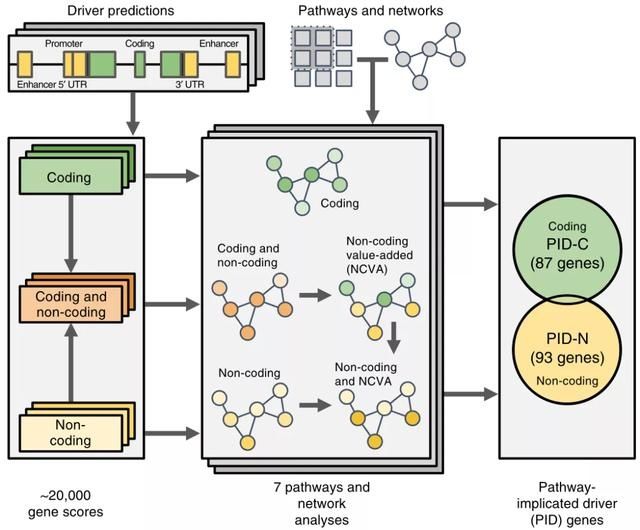
文章图片
▲ Fig.1
“圈”、“联”——交互作用及相关性分析
1.潜在驱动突变的通路和网络分析
接下来,作者就对潜在的驱动突变进行通路和网络分析。他们同时使用了两种通路算法:ActivePathways和一种超几何分析算法——Vazquez;以及五种网络方法:CanIsoNet、Hierarchical HotNet、诱导子网络分析、NBDI22和SSA-ME23。利用以上途径和网络数据库,作者对GS-C、GS-N和GS-CN基因评分运行每种方法,确定了三个相应的基因列表。每种方法对其结果在每个数据集上的统计显著性进行评估。
作者通过网络互作分析观察到显著突变的编码和/或非编码元素之间存在大量交互,这表明通路和网络方法可能有助于优先处理单一元素分析中不显著的罕见驱动事件。这也是这篇研究文献的意义所在。
在具体的通路和驱动分析,以确定驱动基因在长尾的驱动p值编码和非编码突变中,仅使用编码突变可以识别出87个带有编码变异的通路牵连驱动基因(PID-C基因),其中包括68个以前被COSMIC 癌症基因普查(CGC)数据库编目的癌症基因。并且其中的31个基因在PCAWG分析中无统计学意义。说明以上网络分析可以筛选到具有罕见突变的基因,即那些在“长尾”分布的,可能是驱动基因。作者还发现根据CGC数据库对比,这31个FDR > 0.1的基因中有13个是已知的驱动因素。与非PID-C基因相比,PID-C基因的编码基因得分明显较高。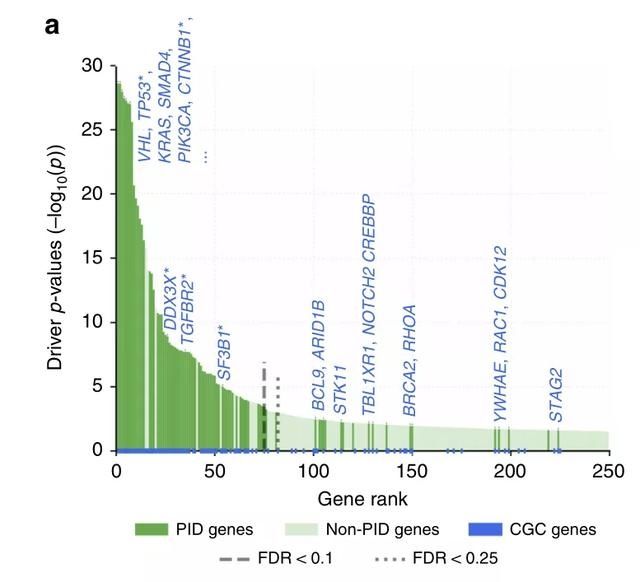
文章图片
▲ Fig.2a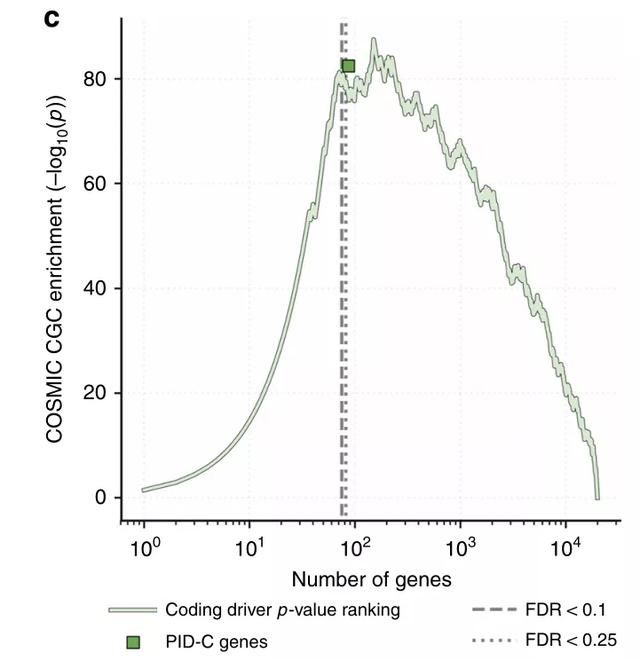
文章图片
▲ Fig.2c
仅使用非编码突变,作者确定了62个基因,发现编码突变中较强的信号在编码和非编码突变中占主导地位。因为非编码突变检测的敏感性较差,作者团队设计了一种“非编码增值”(NCVA)程序。该程序询问了编码突变是否增强潜在非编码驱动基因的发现率,而非只有非编码突变时才被检测到。于是又识别了另外31个基因,与62个单基因非编码突变合并后得到93个带有非编码突变的通路牵连驱动基因(PID-N)。包括了19个先前根据COSMIC癌症基因普查(CGC)数据库确定的癌症基因,显著高于预期的基因。在排除PCAWG分析中具有个体显著非编码元件的8个基因,19个基因同时为PID-N基因和CGC基因,显著富集于预期的基因。
这表明,非编码突变可能会改变某些样本的基因,使其具有周期性编码或结构变异。PID-N基因的非编码基因得分显著高于非PID-N基因。绝大多数PID-N基因与PCAWG驱动因素和功能解释工作组分析不同(图2b),只有三个基因相同:TERT, HES1和TOB1。在这三个基因中,只有TERT在CGC数据库中被记录为已知的癌症基因。因此,作者对通路和网络分析的共识程序显著增强了PCAWG驱动因素和功能解释工作组结果中的显著突变因素。
文章图片
▲ Fig.2b
文章图片
▲ Fig.2d
总的来说,PID-C和PID-N基因在PCAWG驱动因素和功能解释工作组分析中发现了另外121个基因,包括90个新的可能的非编码驱动因素。此外,绝大多数PID-N基因与PID-C基因不同,仅有5个基因相同:CTNNB1、DDX3X、SF3B1、TGFBR2、TP53。虽然这表明编码和非编码驱动突变发生在很大程度上不同的癌症基因集合中,但这两种类型的突变影响着许多相同的标志性癌症进程背后的基因。
2.非编码突变对基因表达的影响
非编码突变可能通过改变转录因子结合位点或其他类型的调控位点发挥作用。
因此,作者评估了PID-N基因的非编码突变是否与同一基因的cis表达变化有关。我们发现,在可以利用RNA-Seq数据检测的90个PID-N基因中,有5个(FDR < 0.3)的cis表达相关性显著(图3)。发现其他四个PID-N基因:TP53、TLE4、TCF4和DUSP22的cis表达相关性显著(图3、补充图10)。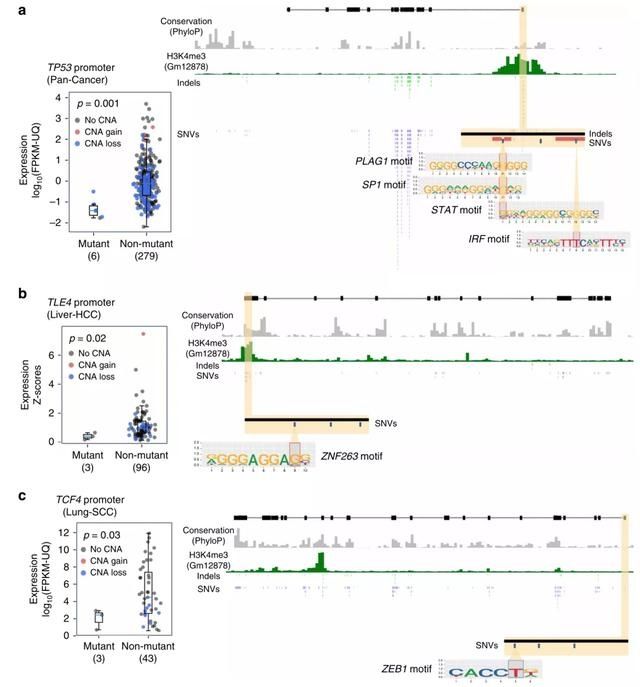
文章图片
▲ Fig.3
接下来,作者对6种不同肿瘤类型的TP53启动子突变的6个肿瘤进行分析,TP53表达明显降低(图3a)。TP53启动子甲基化和表达之间的联系之前已经被研究过,突变样本的减少表达与TP53作为肿瘤抑制基因的众所周知的作用是一致的。PCAWG驱动因素和功能解释的研究也描述了一致的表达变化。TLE4是一种转录辅助抑制因子,在三个具有TLE4启动子突变的肝-肝癌肿瘤中分析,TLE4表达明显减少(图3b)。在三个TCF4启动子突变的肺鳞癌中,TCF4显著低表达(图3c)。DUSP22编码一种磷酸酶信号蛋白,最近被认为是淋巴细胞中的肿瘤抑制因子,DUSP22表达同样显著降低。在5例肺腺瘤患者中,存在DUSP22 3′UTR突变且明显过表达;在三个肺腺瘤患者中存在DUSP22 5 ' UTR突变,然而这些UTR突变是相互排斥的。
推荐阅读
- 山东|探索青年学生助力乡村振兴新模式,2020年“清华学子山东行”圆满收官
- |《我的女友是机器人》首曝花絮,辛芷蕾包贝尔搞怪
- 【地评线】安全开学,精细化防控
- 【地评线】京彩好评:改革不停顿开放不止步是最好的纪念
- 【地评线】安全开学,精细化防控|【地评线】安全开学,精细化防控
- 外交部回应安倍晋三宣布辞职:祝愿他早日康复
- 标准地图|2020年标准地图发布, 一点都不能错!
- 2020|青岛银行公布2020年中期业绩: 资产总额超4405亿元 持续稳健增长
- 2020|扩散周知!自然资源部:2020年标准地图发布
- 外交部|外交部回应安倍晋三宣布辞职:祝愿他早日康复








![[俄罗斯观察者]油价下跌将导致危机,萨卡什维利预测:乌克兰政府不会治理国家](https://imgcdn.toutiaoyule.com/20200322/20200322164237960412a_t.jpeg)









